El proyecto de la Enciclopedia del ADN, llamado ENCODE por Encyclopedia of DNA Elements,
ha estudiado con sumo detalle el ADN humano, tanto la parte codificante
(genoma), como la no codificante (mal llamada hace una década como “ADN
basura”). El hallazgo más notable de este estudio es que el 80% de todo
el ADN contiene elementos vinculados a funciones bioquímicas (es decir,
tiene “actividad bioquímica específica”), desterrando la idea de que
gran parte del ADN es simplemente “basura” evolutiva. Un resultado que
fue anticipado en 2007 cuando este proyecto publicó su análisis del 1%
del ADN y que obliga a redefinir el concepto de gen como unidad mínima
heredada. Gran parte del ADN no codificante, que no se expresa en
proteínas, tiene funciones de regulación, por lo que pueden estar
relacionados con enfermedades y pueden ser dianas terapéuticas.
El ADN humano tiene unos 3.000 millones de bases (“letras” A, G, T, o
C), pero solo el 1% contiene unos 21.000 genes que codifican unas
90.000 proteínas. En el ADN entre los genes, el proyecto ENCODE ha
descubierto unas 70.000 regiones “promotoras” que se ligan a proteínas
para controlar la expresión de los genes. También ha descubierto unas
400.000 regiones “potenciadoras” que regulan (potencian o reducen) la
expresión de genes, incluso de genes muy distantes entre sí. Además, ha
descubierto 2,9 millones de regiones a las que se ligan proteínas (por
ejemplo, factores de transcripción) en los 125 tipos de células
estudiados, de las que unos dos tercios se han descubierto en un solo
tipo celular y no aparecen en ningún otro tipo. De hecho, solo unas
3,700 de estas regiones son compartidas por todas las células.
En el proyecto ENCODE han trabajado unos 442 científicos durante unos
10 años, estudiando mediante 24 tipos de experimentos diferentes un
pequeño trozo de ADN en 147 tipos de células humanas diferentes. El
resultado se publica hoy en una serie de 30 artículos en diferentes
revistas científicas, destacando 6 artículos en Nature. Nos lo cuentan
Joseph R. Ecker, Wendy A. Bickmore, Inês Barroso, Jonathan K. Pritchard,
Yoav Gilad, Eran Segal, “Genomics: ENCODE explained,” Nature 489: 52–55, 06 September 2012, y Ed Yong, “ENCODE: the rough guide to the human genome,” Discover Magazine, 5 Sep. 2012.
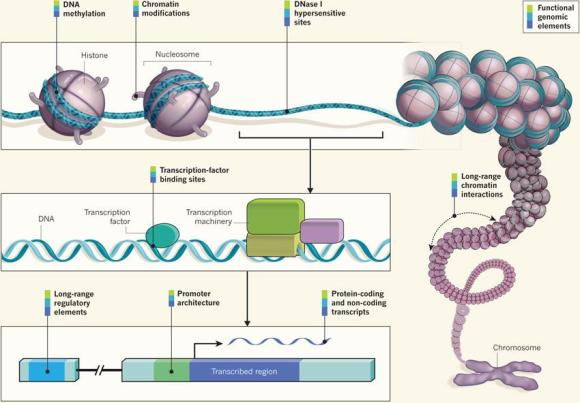
El artículo técnico en Nature que describe en detalle el proyecto
ENCODE es The ENCODE Project Consortium, “An integrated encyclopedia of
DNA elements in the human genome,” Nature 489: 57–74, 06 September 2012. También es interesante el artículo de Ewan Birney, “The making of ENCODE: Lessons for big-data projects,” Nature 489: 49–51, 06 September 2012,
del que he extraído la siguiente figura. Por cierto, en 2007 ya se
publicaron conclusiones similares en Nature cuando se estudió el 1% del
ADN, en concreto, en The ENCODE Project Consortium, “Identification and
analysis of functional elements in 1% of the human genome by the ENCODE
pilot project,” Nature 447: 799-816, 14 June 2007.

GENCODE es la parte del proyecto ENCODE que está catalogando los
trozos de ARN que se transcriben a partir del ADN, que constituyen casi
el 75% de todo el ADN. Sus resultados exigen una nueva definición del
concepto de gen. El artículo técnico es Sarah Djebali et al., “Landscape
of transcription in human cells,” Nature 489: 101–108, 06 September 2012. Los
sitios hipersensibles a la ADNasa I (DHSs por DNase I hypersensitive
sites) son marcadores en la cromatina para la regulación del ADN,
incluyendo potenciadores (enhancers), promotores, aisladores (insulators), silenciadores, regiones de control (locus control regions)
y factores de transcripción. En la región del ADN estudiada se ha
duplicado el número de DHSs conocidos. Los artículos técnicos son Robert
E. Thurman et al., “The accessible chromatin landscape of the human
genome,” Nature 489: 75–82, 06 September 2012, y Shane Neph et al., “An expansive human regulatory lexicon encoded in transcription factor footprints,” Nature 489: 83–90, 06 September 2012. La
red que interconecta los factores de transcripción es estudiada en Mark
B. Gerstein et al., “Architecture of the human regulatory network
derived from ENCODE data,” Nature 489: 91–100, 06 September 2012.
La red que conecta los genes y la parte no codificante del ADN que
regula su expresión es muy compleja, contradiciendo el dogma de que la
regulación de un gen se realiza mediante elementos reguladores próximos
en la cadena de ADN. El artículo técnico es Amartya Sanyal et al., “The
long-range interaction landscape of gene promoters,” Nature 489: 109–113, 06 September 2012.
Resumiendo mucho, lo que nos muestra el proyecto ENCODE es que el ADN
y la regulación bioquímica de la célula es mucho más compleja de lo que
nunca pudimos imaginar. Una de las cosas que más me interesan sobre el
ADN, el estudio detallado de los aspectos dinámicos de la regulación
génica, está más allá de los objetivos del proyecto ENCODE y requiere el
desarrollo de nuevas tecnológicas. Serán necesarias muchas décadas para
que podamos empezar a entender el funcionamiento complejo de cada
célula humana a partir de su ADN y su epigenoma.
PS: Claudio nos recomienda en los comentarios la lectura de Ewan Birney (ENCODE Project), “ENCODE: My own thoughts,” Ewan’s Blog; Bioinformatician at Large, 5 Sept. 2012.
¿Qué significa que el 80% del genoma tiene una función? Que
tiene actividad bioquímica específica; un elemento del ADN es
“funcional” si cambia alguna propiedad bioquímica de la célula. Solo el
8% del ADN está en contacto con proteínas (como los sitios de unión a
los factores de transcripción); incluso, si se incluyen los exones, este
porcentaje solo sube al 9%. Por ello hay que tener cuidado a la hora de
interpretar la definición de “funcional” asociada al 80% del ADN.
¿Qué significa este 80% desde el punto de vista de la evolución? Todo
el ADN que sufrido el efecto de la selección en la parte más reciente
de la evolución humana es considerado “funcional,” incluso si aún no
sabemos si tiene efecto fenotípico o no. Solo entre el 10% y el 20% se
espera que tengan dicho efecto. Dentro de la colaboración ENCODE se han
tenido debates muy intensos sobre qué vocabulario utilizar. Al final la
decisión de consenso que se ha tomado puede ser discutible pero está
bien fundamentada en los artículos.
PS: Vídeo de Nature sobre el proyecto ENCODE.
También recomiendo la presentación Flash de Nature ENCODE explorer.
PS (7 sep 2012): Recomiendo ver la
entrevista en vídeo a Lluís Montoliu, investigador del CNB (Centro
Nacional de Biotecnología) “Cinco respuestas sobre el proyecto ENCODE y el “ADN oscuro”,” realizada por Antonio Martínez Ron @aberron y Miguel Fernández Flores para lainformacion.com.
PS (8 sep 2012): Recomiendo encarecidamente, porque me ha encantado, la lectura de la entrada de PaleoFreak, “FAQ: El Proyecto ENCODE y el supuesto fin del ADN basura,” 7 sep. 2012. En especial destaco que “¿Han dicho los científicos del ENCODE que sus hallazgos refutan la existencia del ADN basura? Creo que no, aunque no descarto que lo hayan dicho en entrevistas. Ni siquiera encuentro la palabra junk en los trabajos de libre acceso publicados en Nature.” Lo que es cierto en los seis artículos técnicos de ENCODE publicados en Nature, no así en los artículos sobre ENCODE publicados en dicha revista.
Tomado de: http://francisthemulenews.wordpress.com/2012/09/06/el-proyecto-piloto-encode-dice-adios-al-adn-basura-el-80-del-adn-tiene-funciones-bioquimicas/





No hay comentarios:
Publicar un comentario